- ·
- 【circRNA测序】 circRNA-seq
- 产品介绍
- 案例解析
- 结果展示
- 送样建议
- FAQ
利用circRNA对RNase R的耐受性从而富集circRNA,去rRNA后进行链特异建库,采用Illumina平台对circRNA文库进行测序,通过生信分析得到circRNA的表达结果。
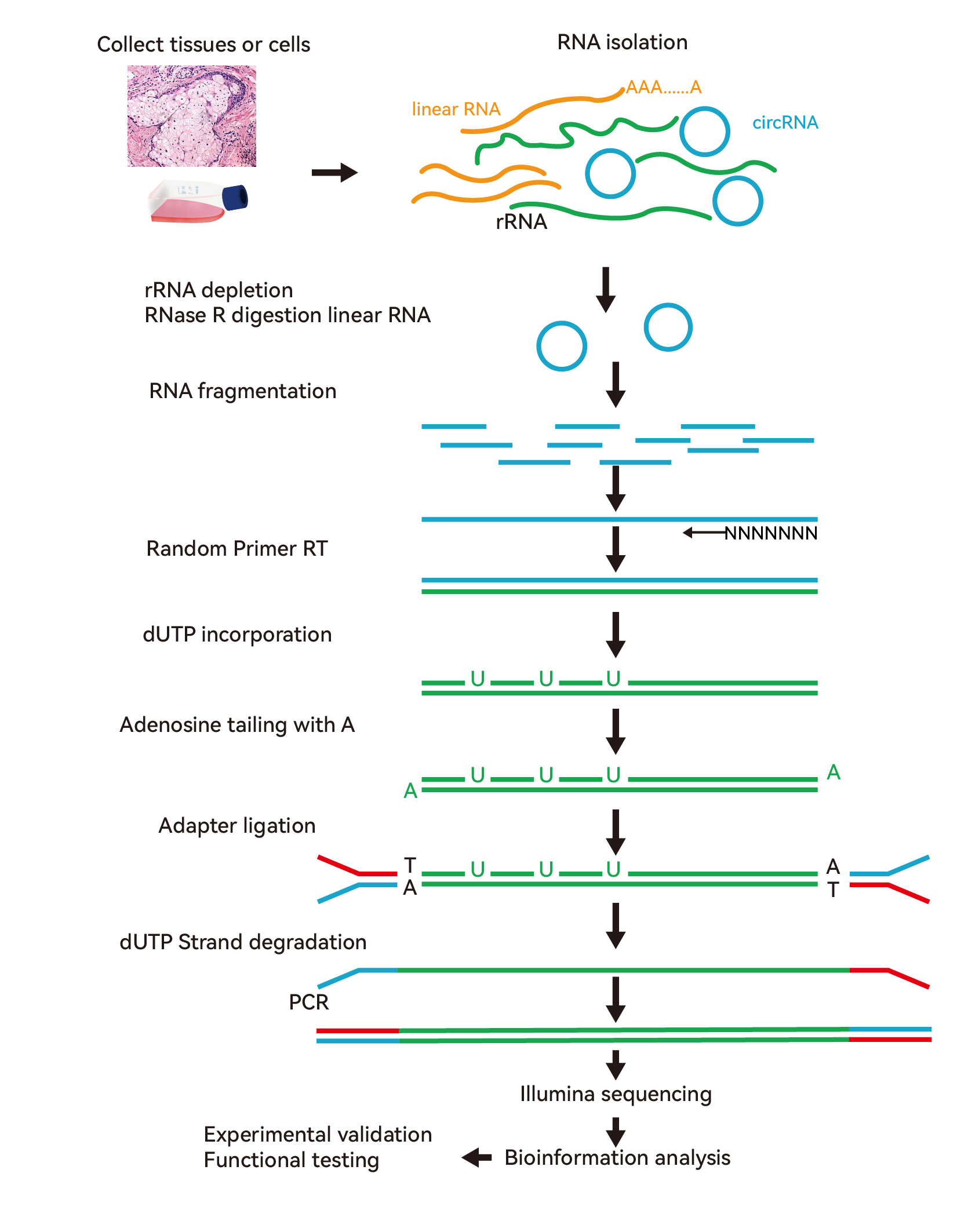
circRNA测序技术路线
测序方案
测序平台:Illumina Novaseq 6000/NovaSeq X Plus
测序模式:PE150
测序数据量:10 Gb raw data
人喉鳞状细胞癌中circRNA的RNA-seq表达谱
RNA-Seq profiling of circular RNAs in human laryngeal squamous cell carcinomas
Lu et al. Molecular Cancer (2018) 17:86(IF:6.204)
测序策略:circRNA测序(RNase R+)
样本分组:10例,5例肿瘤组织(2例高分化,3例中分化),5例正常组织
人喉鳞状细胞癌(LSCC)是起源于喉黏膜上皮组织的恶性肿瘤,是头颈部肿瘤中恶性程度非常强的肿瘤,世界范围内每年发生率占比2.4%,近年内还有逐渐增加趋势。本研究主要关注circRNA在LSCC疾病中的表达情况。研究者选择10例LSCC患者临床组织标本(2例高分化、3例为中分化、 5例正常)进行RNA高通量测序,分析样本中circRNA表达情况。共鉴定出21444个circRNA,其中在LSCC样本中显著上调的circRNA 29个,显著下调circRNA 19个,进一步结合临床特征(高分化,中分化和正常)进行分类,将差异表达circRNA缩小到18个和5个(图1)。CircRNA-miRNA-mRNA调控网络预测分析,发现20个失调控circRNA与多种肿瘤相关的miRNA相关,信号通路KEGG富集分析发现,过氧化物酶体PPAR、轴突导向、Wnt和细胞周期通路与LSCC密切相关(图2)。随后的qPCR验证结果显示(图3),has_circ:chr20:31876585-31,897,648可以很好区分LSCC标本和正常标本,表明circRNA可作为LSCC潜在的理想标志物。
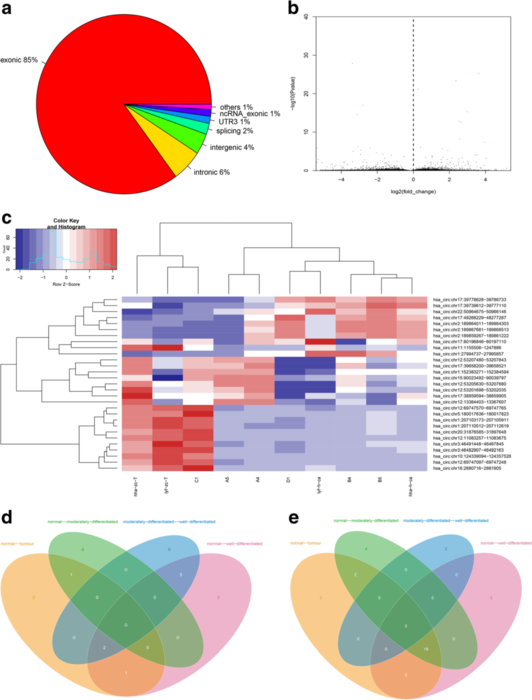 图1 LSCC样本中circRNA差异表达谱
图1 LSCC样本中circRNA差异表达谱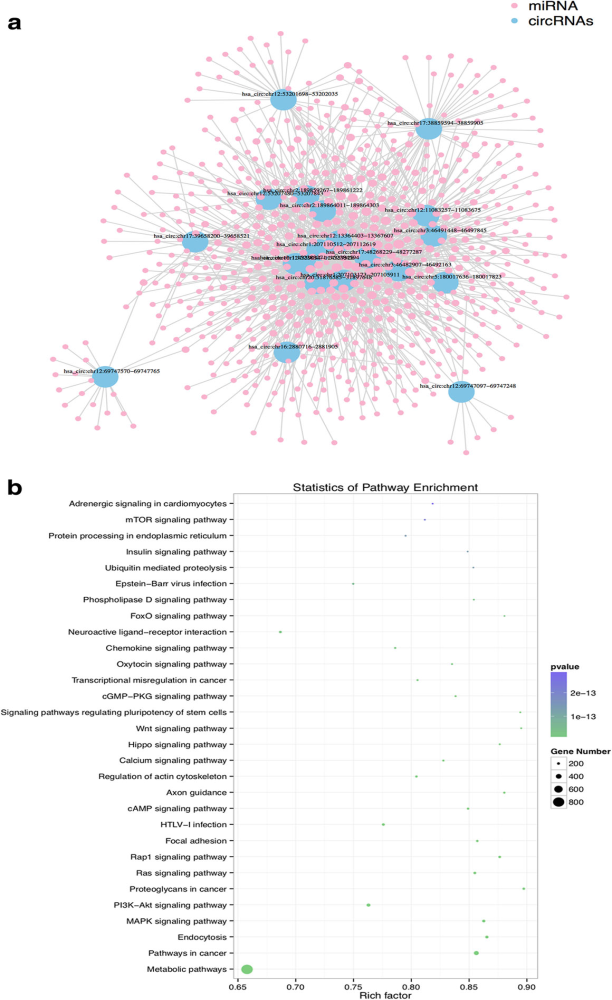 图2 CircRNA及靶向miRNA网络调控图与KEGG代谢通路富集分析
图2 CircRNA及靶向miRNA网络调控图与KEGG代谢通路富集分析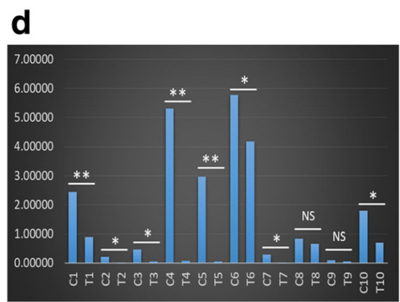
图3 hsa_circ:chr20:31876585–31,897,648在10对LSCC肿瘤样本及正常样本中的Q-PCR验证
生物信息分析
基础分析
原始数据质控检查
比对结果质控检查
基因覆盖度分析
circRNA预测及鉴定
circRNA序列预测
基于circRNA表达的样品主成分分析
高级分析
circRNA差异分析
差异circRNA宿主基因的GO功能分析
差异circRNA宿主基因的KEGG通路分析
差异circRNA宿主基因的Rectome通路分析
差异circRNA的靶向miRNA预测分析
差异circRNA及靶向miRNA网络调控制图
部分结果展示
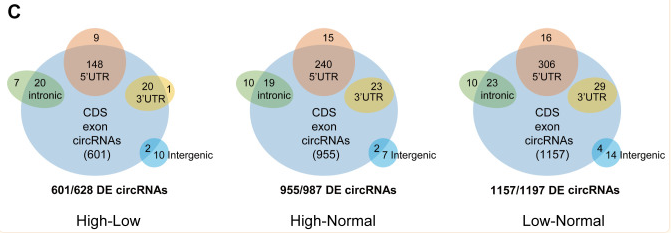
图1:根据不同的基因组位点(外显子、内含子、反义、基因内和基因间)对表达差异显著的circRNA进行分类
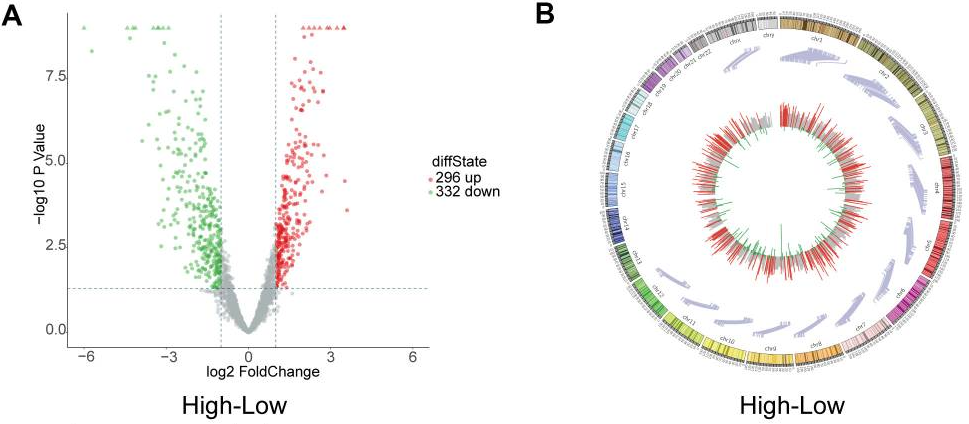
图2:绘制火山图、Circos图揭示每个circRNA在人类染色体上的位置
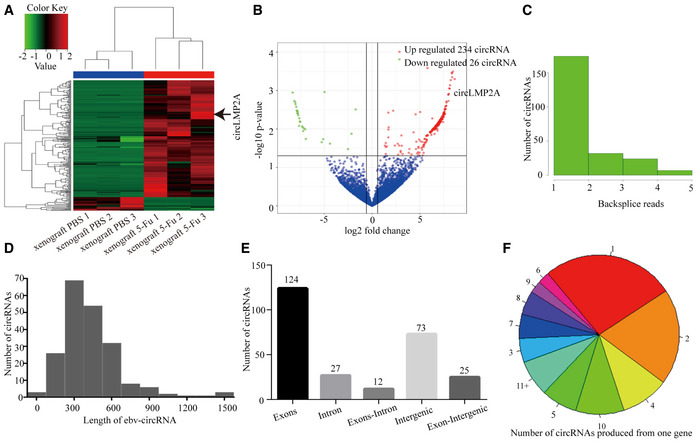
图3:热图、火山图分析circRNA的表达差异倍数
Table.circRNA-seq原始样本送样建议
送样类型
送样量
备注
细胞 (细胞数)
≥1*10^6
无支原体污染
动物组织
≥100mg
植物组织
≥100mg
全血
≥4mL
FFPE
8-10片,未染色,厚度10μm
Table.circRNA-seq RNA样本送样建议
送样类型
送样量
完整性(RIN值)
浓度
其他
纯度
total RNA(组织、细胞、全血等)
≥4ug
≥7
≥100ng/μl
无DNA,蛋白/盐离子等污染,样本无色透明不粘稠
FFPE组织RNA
≥4ug
≥3
/
DV200>30%
*更具体的送样方法请咨询销售或技术支持
物种范围:人、小鼠、大鼠等哺乳动物,拟南芥、水稻、番茄、玉米、大豆等植物,其他物种详询销售或技术支持
Q1:circRNA测序与全转录组测序有什么不同?
A1:全转录组测序采用去rRNA链特异性的建库方式,数据分析内容包含了mRNA、lncRNA及circRNA全部信息,数据质量较好。CircRNA测序采用去rRNA去线性链特异性的建库方式,通过RNase R 消化线性RNA从而富集circRNA,对建库起始量要求较高(一般大于2μg),测序数据质量较全转录组稍差,但可将circular junction reads数提高10倍以上,更适合专注circRNA研究的项目。

 购物车
购物车



 广州市黄埔区开源大道11号科技企业加速器A区6栋2楼
广州市黄埔区开源大道11号科技企业加速器A区6栋2楼


 geneseed@geneseed.com.cn
geneseed@geneseed.com.cn