
- 首页
- >
- GENESEED
- >
翻译组研究解决方案

根据中心法则,DNA是遗传信息的载体,蛋白质是生物活动的主要执行者。蛋白表达丰度受RNA合成(转录)、RNA降解(转录后)、蛋白质合成(翻译)和降解(翻译后)等多个过程的影响。
RNA翻译蛋白过程连接转录组和蛋白质组,极消耗能量,因而受严密的调控,以节省细胞资源,并避免毒性蛋白质的产生。
翻译速率可决定蛋白质的折叠构象、定位与功能、产量及对应的细胞表型。蛋白质合成(翻译)速率与最终蛋白质丰度的相关性较高,翻译速率对蛋白质水平的贡献最大。因此,翻译组研究是分析基因表达和调控水平的重要环节。

更重要的是,近年来陆续有研究发现,曾被认为非编码的RNA(ncRNA)可编码或翻译蛋白,而翻译组分析可精准可视化研究编码全新隐秘多肽的ncRNA(如circRNA、lncRNA)。

01丨多聚核糖体图谱分析(Polysome profiling)
——传统的“黄金标准”
Polysome profiling技术是基于蔗糖浓度梯度溶液超离心,将细胞质RNA分离成多个组分:游离RNA,结合核糖体40S或60 S亚基、单核糖体(80S)或多聚核糖体等。一条翻译的mRNA可同时结合多个核糖体,结合核糖体组分的RNA与总胞质RNA的比例可以间接反映翻译水平。
丨Polysome profiling步骤
①裂解细胞:添加翻译延伸抑制剂,在增殖阶段裂解细胞,分离细胞质裂解液;
②超速离心:把裂解液转移到梯度蔗糖溶液后,超速离心将RNA分离成不同的组分;
③收集组分:利用密度梯度分馏系统,UV吸光度检测核酸浓度并收集各个组分;
④回收产物:从蔗糖溶液组分中回收RNA复合物;
⑤分析产物:绘制核糖体图谱;RT-qPCR分析特定RNA;高通量测序分析全局RNA概况;Western Blot或质谱分析蛋白质。
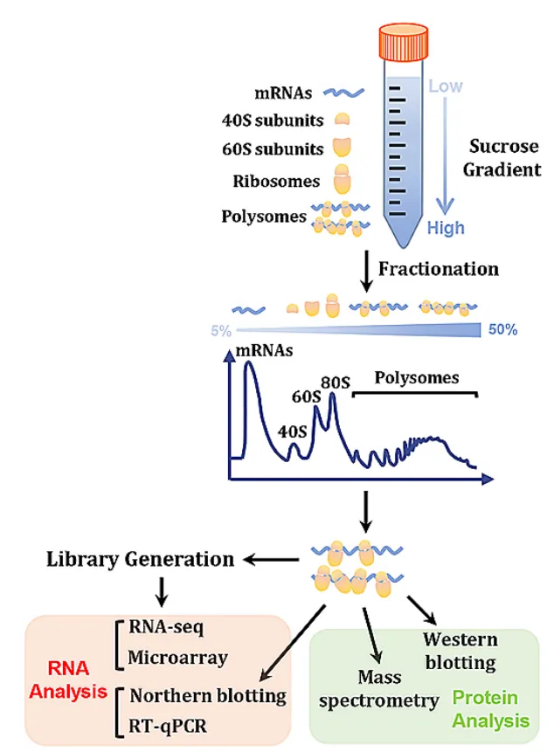
图1 Polysome Profiling技术路线图。(source:Su D, et al., 2024)
丨Polysome profiling技术优势
① 直观反映细胞和组织内的翻译状态;
② 对胞质RNA翻译水平分级,可针对感兴趣组分进一步分析不同的翻译机制;
③ 结合高通量测序可全面、精准、高通量反映翻译组概况。
Polysome profiling技术局限
① 需专门设备在常规实验室可能无法获得;
② 操作较繁琐:需一系列繁琐的溶液配置、离心、分离、检测等操作步骤,耗时长且对操作者要求较高;
③ 容易受干扰:细胞内普遍存在高分子量的复合物,容易对多聚核糖体收集和检测造成干扰。
——更详细地分析翻译的强大工具
Ribo-seq可获得核糖体沿着翻译mRNA的精确位置,从而得到详细翻译事件的直接证据,可在密码子分辨率上进行全翻译组检测。
丨Ribo-seq步骤
① 使用延长抑制剂阻止核糖体移位,然后收取细胞;
② 用RNase消化细胞裂解物,裸露的RNA片段打断,而核糖体保护RNA片段得以保留;
③ 去除干扰的rRNA,去除核糖体,收集核糖体保护片段(RPF),也称为核糖体足迹;
④ 建库,针对RPF深度测序并分析获取的核糖体足迹数据。
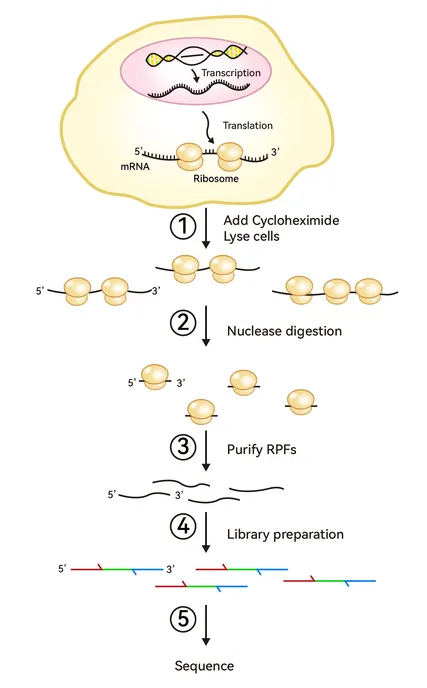
丨Ribo-seq技术优势
① 提供精确而丰富的核糖体位置信息,可更详细分析翻译调控事件,包括翻译起始、延伸、停止和终止或非常规的翻译事件,如非AUG介导的翻译起始、停止密码子读取、以及翻译新的小开放阅读框(sORF),或以前被认为是非编码RNA (ncRNAs)的非典型可翻译RNA;
② 可瞬时捕获翻译过程的状态,定量分析翻译效率,对研究翻译调控动态变化具有重要意义;
③ Ribo-seq可同时捕获大量基因的翻译活动,为构建全局性翻译提供可能,结合RNA-seq,可系统研究基因表达与翻译调控之间的关系。
丨Ribo-seq技术局限
① 细胞中不同的mRNA的核糖体延伸率不同,无法区分翻译活跃的核糖体和停滞状态的核糖体,可能导致翻译效率的偏差;
② 小RNA片段(如调控性非编码RNA)的污染可能导致翻译组数据的误解;
③ 核糖体的构象、不适当的核酸酶酶切和特定的RNA二级结构,都可能影响足迹长度。
03丨核糖体-新生体-链复合体测序(RNC-seq)
——全长翻译mRNA测序
RNC-seq技术分离出核糖体结合RNA进行高通量测序,不经过RNase处理,保留RNA全长信息。
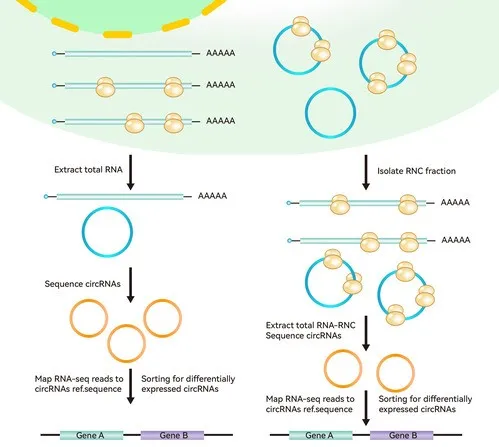
丨RNC-seq技术优势
① 实验操作较简便;
② 较长的片段建库,排除了潜在的污染物(如调控性小RNA);
③ 长片段更可能跨越mRNA和circRNA的剪接位点,检测更多的选择性剪接(AS)异构体或翻译circRNA;
④ 与RNA-seq结合使用,可通过计算翻译比率,评估哪些mRNA剪接变体在全基因组范围内翻译。
丨RNC-seq技术局限
①RNC RNA易降解;
②一个或多个核糖体结合的翻译RNA分子被统一分析,而不是像polysome Profiling分成多个组分进行分析,翻译效率分析准确性较低;
③若无合适计算分析,RNC-seq由于丢失了翻译RNA上核糖体的位置信息,而无法精确定义非常规ORF的位置,只能为非规范ORF的翻译提供间接证据。
——翻译调控研究新技术
停滞的核糖体(stalled ribosomes)是指在翻译过程中暂时停止或显著放慢移动的核糖体。核糖体停滞可能由于多种原因,包括罕见密码子的存在、mRNA的超二级结构、损坏的mRNA或特定的结合蛋白质的存在,直接导致异常蛋白的合成或核糖体的缺乏,从而调控翻译。核糖体碰撞形成的串联双核糖体,可介导共翻译事件,对细胞稳态具有重要作用。
Disome-seq是基于Ribo-seq发展的技术,通过分离核糖体-RNA复合物,用RNase进行酶切消化,裸露的RNA被酶切,而核糖体保护的RNA得以保留。然后分离纯化得到双核糖体保护的RNA片段,进行建库测序。
Disome-seq具有Ribo-seq技术的优势的同时,可针对双核糖体碰撞发生的RNA进行分析,有利于研究共翻译等特殊翻译调控机制。
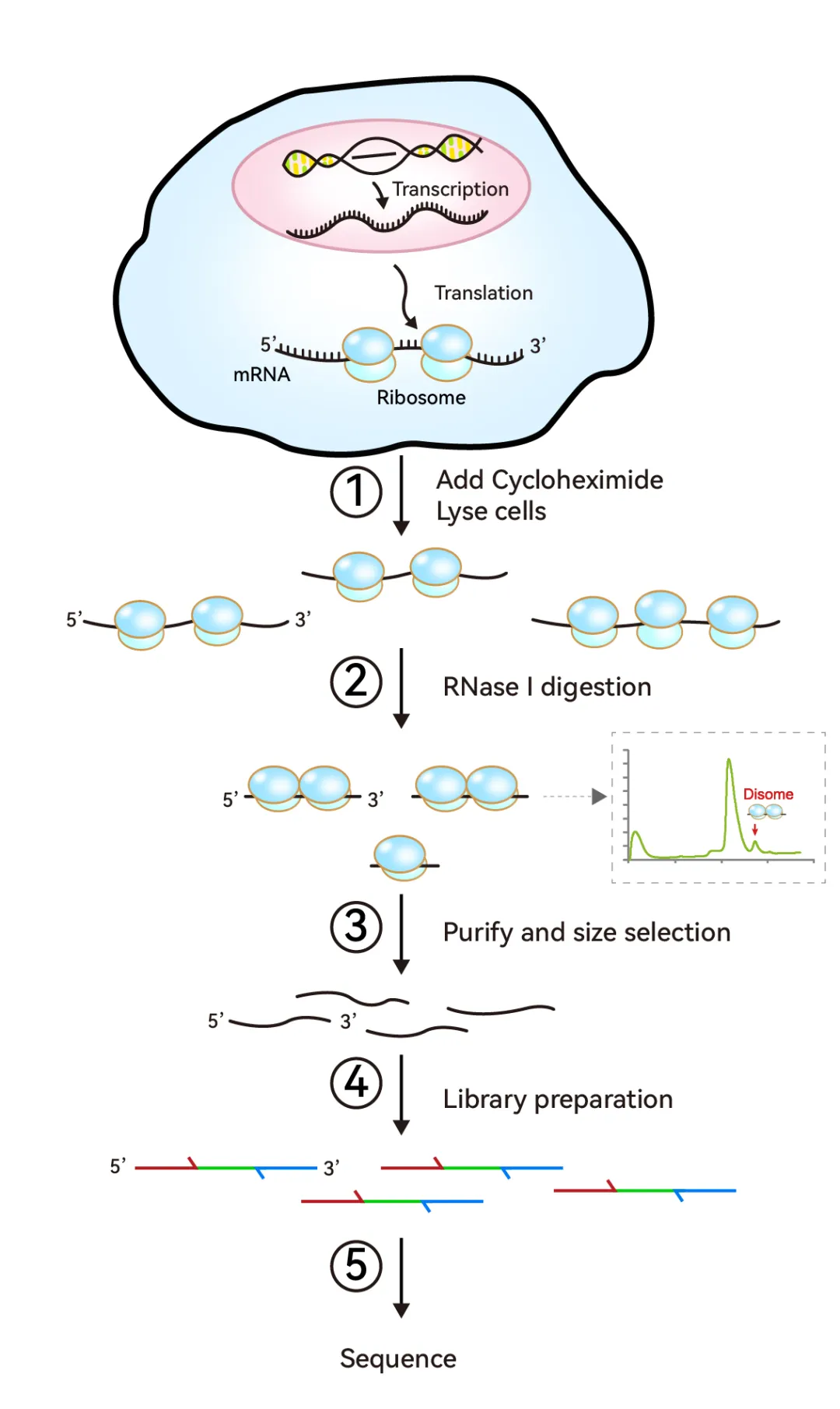
丨Disome-seq技术优势
①揭示全局RNA中碰撞双核糖体的足迹;
②针对性研究翻译停滞、共翻译等特殊的翻译调控事件。
丨Disome-seq技术局限
① 仅针对性分析串联双核糖体结合的RNA;
② 对样品处理和分析的要求较高,可通过增加核糖体图谱分析环节,通过检测双核糖体的特征峰,筛选合格样本进入下游分析流程,有效保障建库测序数据质量。

Polysome profiling可以准确研究翻译效率,研究翻译调控、翻译活性等;
Ribo-seq可以研究RNA的翻译机制、翻译起始和终止位点及ORF等;
RNC-seq可以研究翻译复合物的组成和活性,以及挖掘潜在可翻译的非典型RNA;
Disome-seq可以分析双核糖体或核糖体碰撞事件研究特定的翻译调控机制。
其中,Ribo-seq和RNC-seq实际上是从两个不同的方面评估RNA翻译,两者不能相互替代。
吉赛翻译组技术服务内容
吉赛生物可提供Ploysome profiling、Ribo-seq和RNC-seq一系列经典翻译组研究技术服务,还可提供Polysome-seq和Disome-seq,可根据个性化需求选取不同、单/多个分离组分进行建库测序。翻译组研究无烦恼!
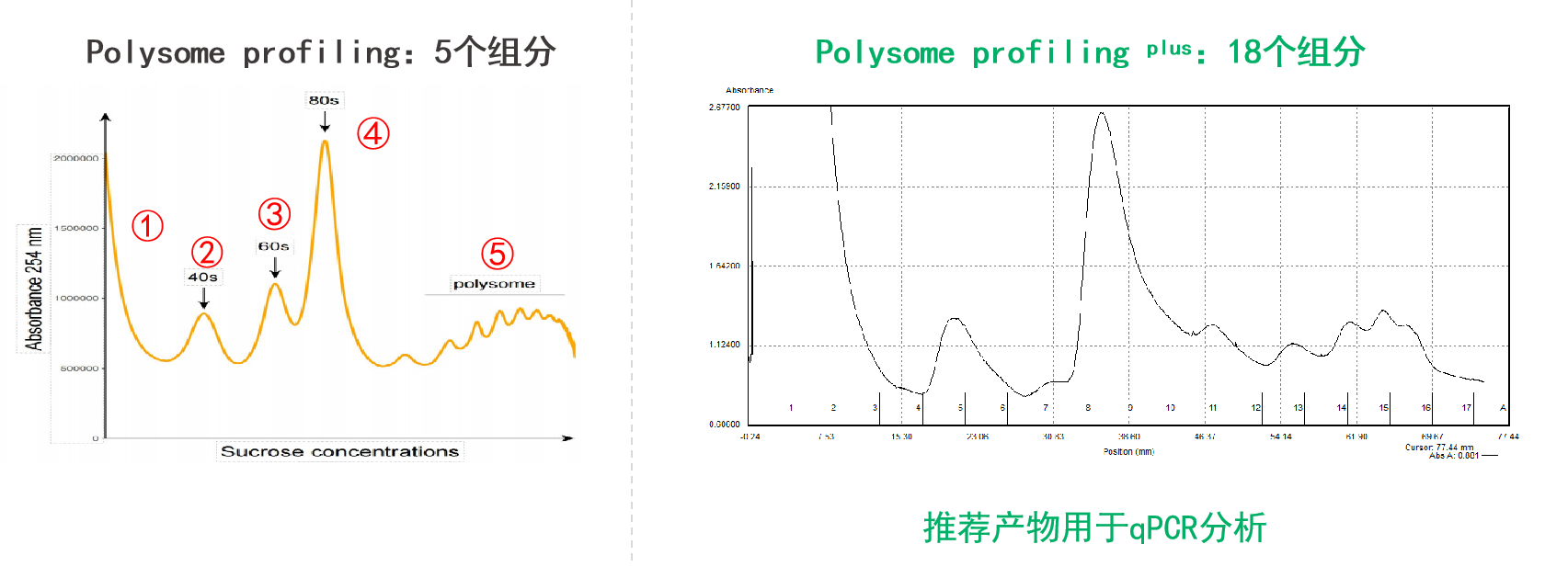
Polysome Profiling图谱示例
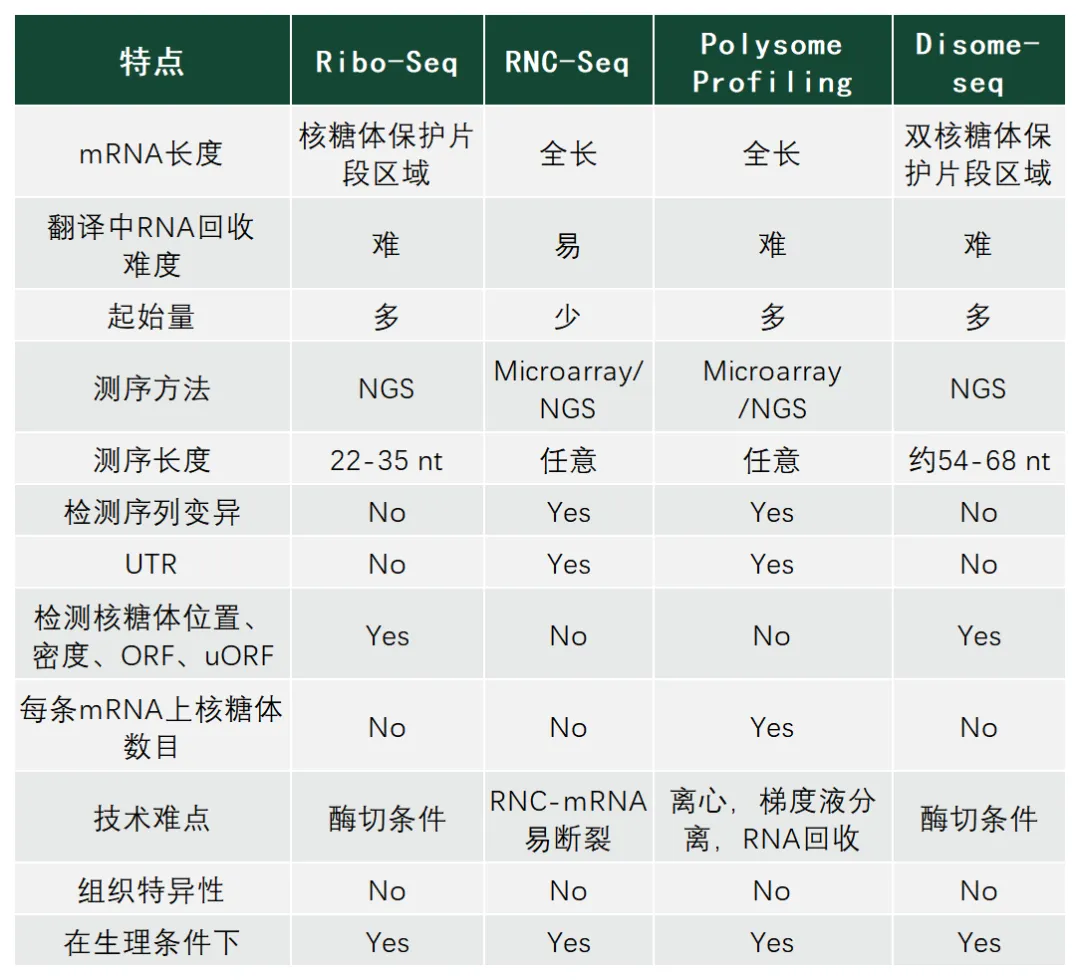
特点
mRNA长度
全长
核糖体保护片段区域
全长
双核糖体保护片段区域
翻译中RNA回收难度
难
难
易
难
起始量
多
多
少
多
测序方法
Microarray/NGS
NGS
Microarray/NGS
NGS
测序长度
任意
22-35 nt
任意
约54-68 nt
检测序列变异
√
×
√
×
UTR
√
×
√
×
检测核糖体位置、密度、ORF、uORF
×
√
×
√
每条mRNA上核糖体数目
√
×
×
×
技术难点
离心,梯度液分离,RNA 回收
酶切条件
RNC-mRNA 易断裂
酶切条件
生理条件
√
√
√
√
吉赛服务项目
图谱(5或18组分)
Ribo-seq测序分析
RNC-seq测序分析
无图谱-Disome-seq测序分析
图谱+RNA(5或18组分)
图谱+qPCR(5或18组分)
图谱+WB(5或18组分)
图谱+Disome-seq测序分析
图谱+测序(组分⑤单文库)
图谱+测序(双文库-Light+Heavy组分)
图谱+测序(双文库-Free+Binding组分)
吉赛生物可提供Ploysome profiling、Ribo-seq和RNC-seq一系列经典翻译组研究技术服务,还可提供Polysome-seq和Disome-seq,可根据个性化需求选取不同、单/多个分离组分进行建库测序。翻译组研究无烦恼!

Polysome Profiling图谱示例
特点 | ||||
mRNA长度 | 全长 | 核糖体保护片段区域 | 全长 | 双核糖体保护片段区域 |
翻译中RNA回收难度 | 难 | 难 | 易 | 难 |
起始量 | 多 | 多 | 少 | 多 |
测序方法 | Microarray/NGS | NGS | Microarray/NGS | NGS |
测序长度 | 任意 | 22-35 nt | 任意 | 约54-68 nt |
检测序列变异 | √ | × | √ | × |
UTR | √ | × | √ | × |
检测核糖体位置、密度、ORF、uORF | × | √ | × | √ |
每条mRNA上核糖体数目 | √ | × | × | × |
技术难点 | 离心,梯度液分离,RNA 回收 | 酶切条件 | RNC-mRNA 易断裂 | 酶切条件 |
生理条件 | √ | √ | √ | √ |
吉赛服务项目 | 图谱(5或18组分) | Ribo-seq测序分析 | RNC-seq测序分析 | 无图谱-Disome-seq测序分析 |
图谱+RNA(5或18组分) | ||||
图谱+qPCR(5或18组分) | ||||
图谱+WB(5或18组分) | 图谱+Disome-seq测序分析 | |||
图谱+测序(组分⑤单文库) | ||||
图谱+测序(双文库-Light+Heavy组分) | ||||
图谱+测序(双文库-Free+Binding组分) |

 购物车
购物车


 广州市黄埔区开源大道11号科技企业加速器A区6栋2楼
广州市黄埔区开源大道11号科技企业加速器A区6栋2楼


 geneseed@geneseed.com.cn
geneseed@geneseed.com.cn