- ·
MeRIP-Seq
- 产品介绍
- 案例解析
- 结果展示
- 送样建议
- FAQ
服务介绍
通过m6A特异性抗体识别并结合具有m6A修饰的RNA片段进行免疫共沉淀,对富集下来的RNA片段进行高通量测序。结合生物信息学分析,即可在全转录组范围内对m6A修饰进行系统研究。
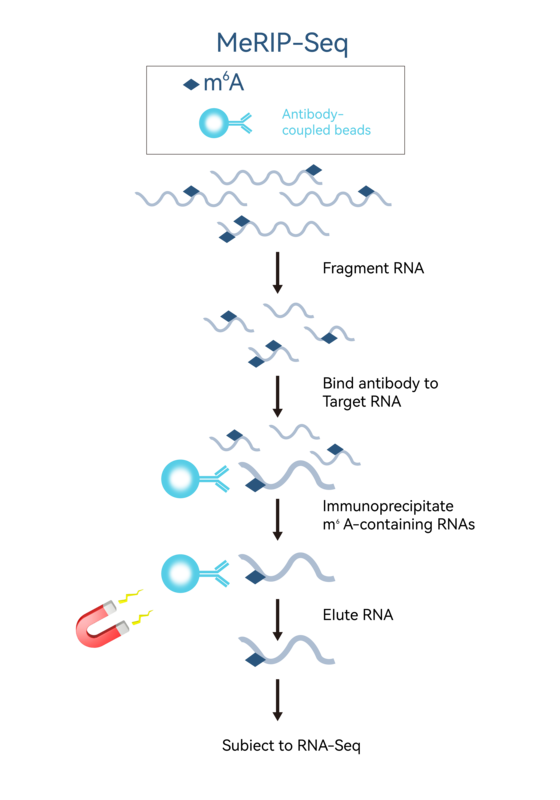
RNA m6A甲基化测序技术路线
测序方案
上机平台:Illumina Novaseq 6000/NovaSeq X Plus
测序模式:PE150
测序数据量:12 Gb raw data案例1
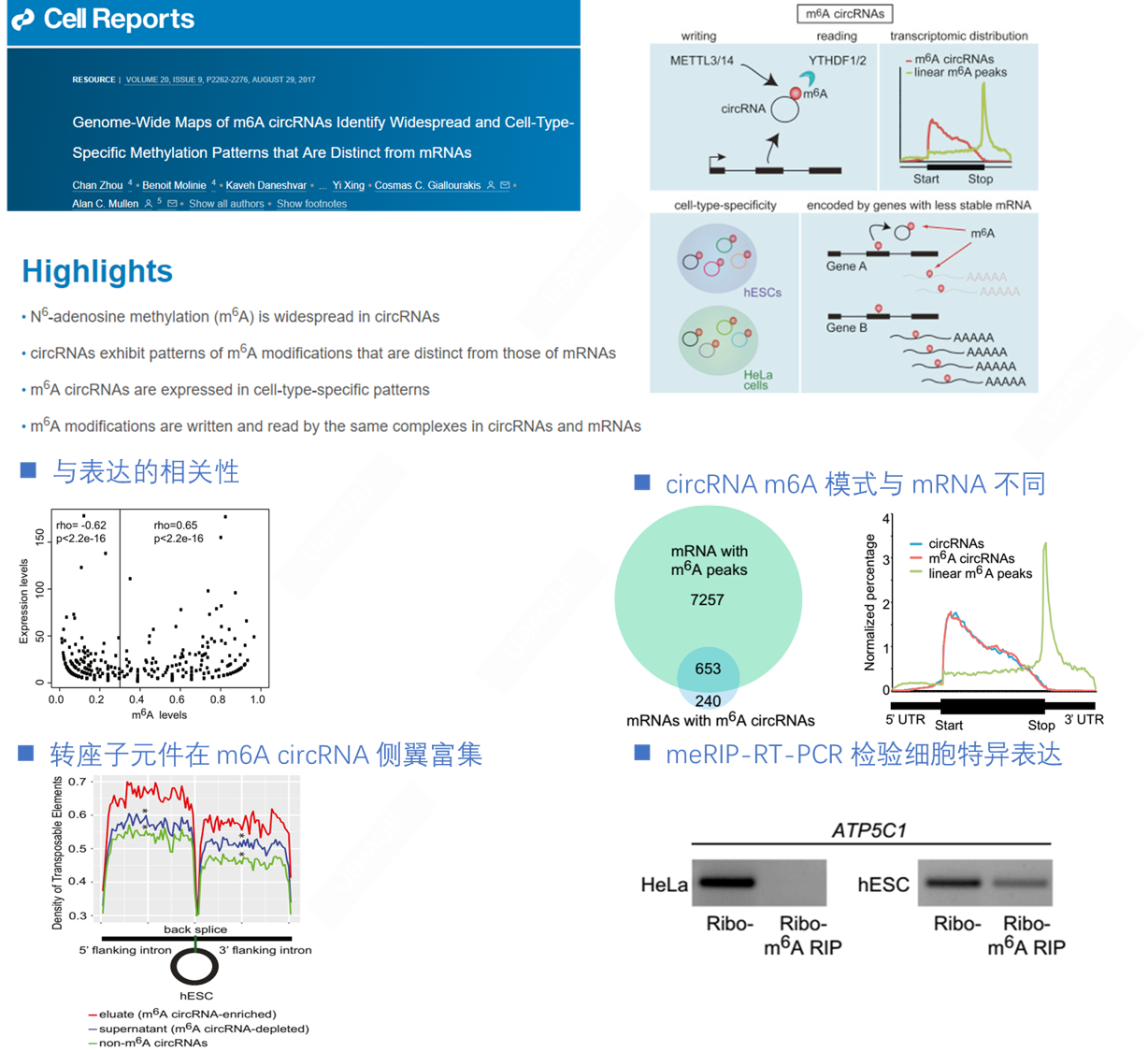
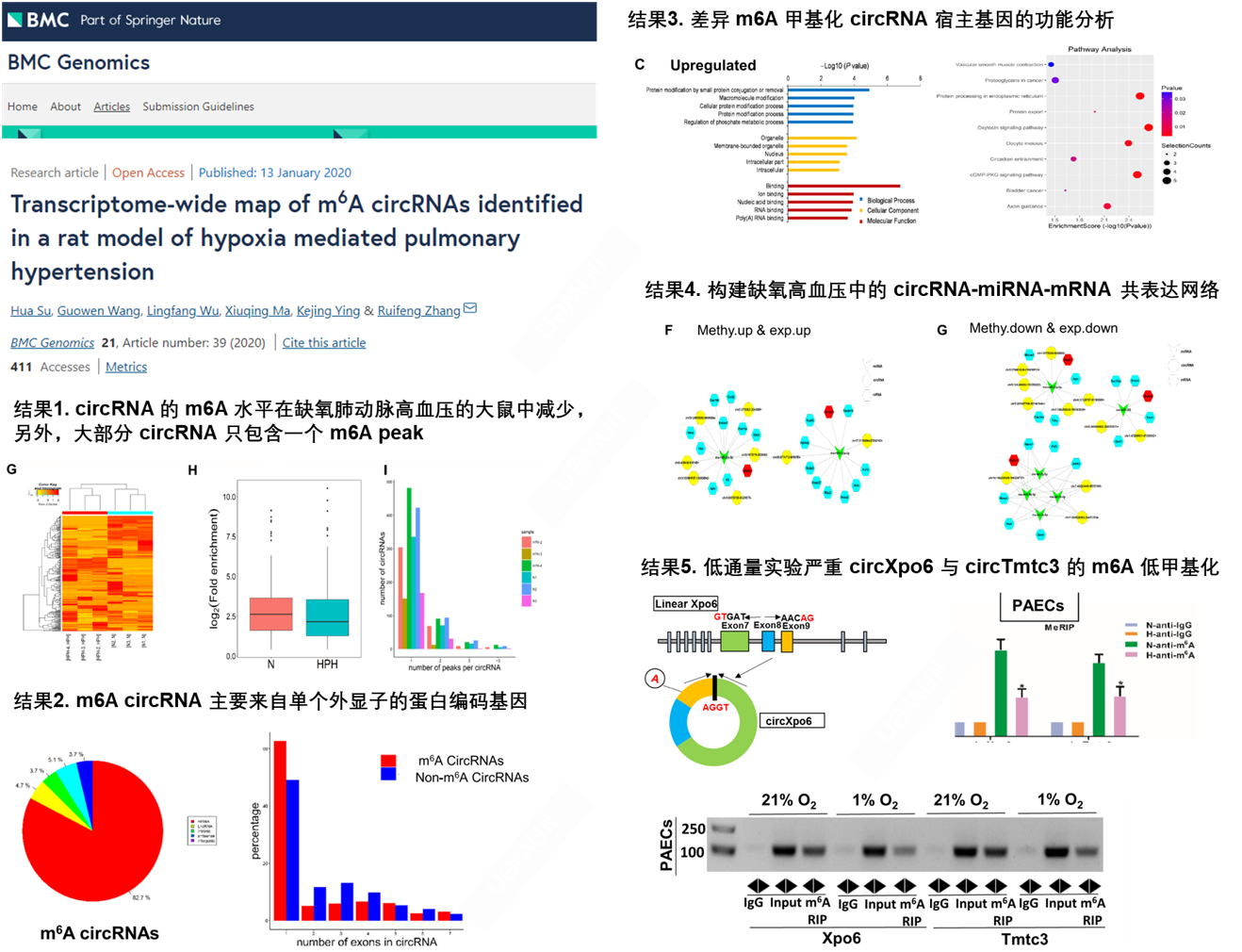
案例2
生物信息分析
基础分析
测序数据质量评估
比对结果质控
基因覆盖度分析
m6A peaks识别
m6A peaks特征注释
m6A mRNA分析
甲基化mRNA的motif分析
m6A lncRNA分析
甲基化lncRNA的motif分析
circRNA识别鉴定分析
circRNA的m6A富集分析
甲基化circRNA的motif分析
高级分析
甲基化基因的差异表达分析
差异甲基化基因GO功能富集分析
差异甲基化基因KEGG通路分析
差异甲基化基因reactome通路分析
甲基化lncRNA的差异表达分析
差异甲基化lncRNA的顺式编码基因的GO功能富集分析
差异甲基化lncRNA的顺式编码基因的KEGG通路分析
差异甲基化lncRNA的顺式编码基因的Reactome通路分析
甲基化circRNA的差异表达分析
差异甲基化circRNA宿主基因的GO功能富集分析
差异甲基化circRNA宿主基因的KEGG通路分析
差异甲基化circRNA宿主基因的Reactome通路分析
个性化分析
与转录组关联分析
关联分析四象限图
部分结果示例

图1. RIP 富集分布分析
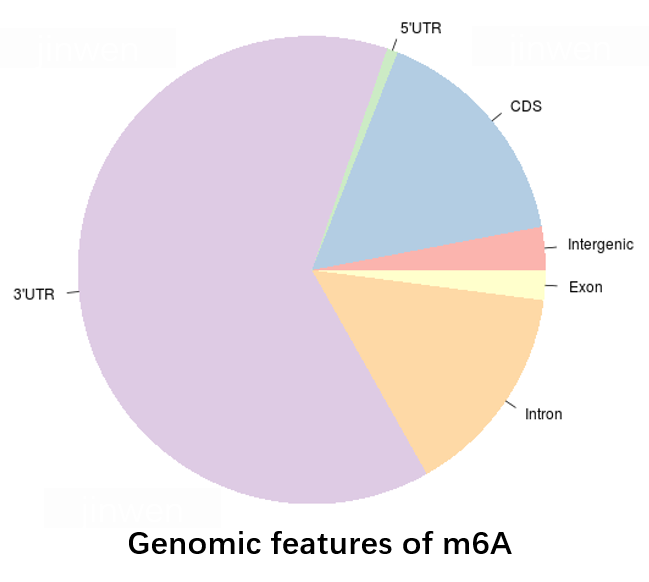
图2. m6A基因组特征分布分析
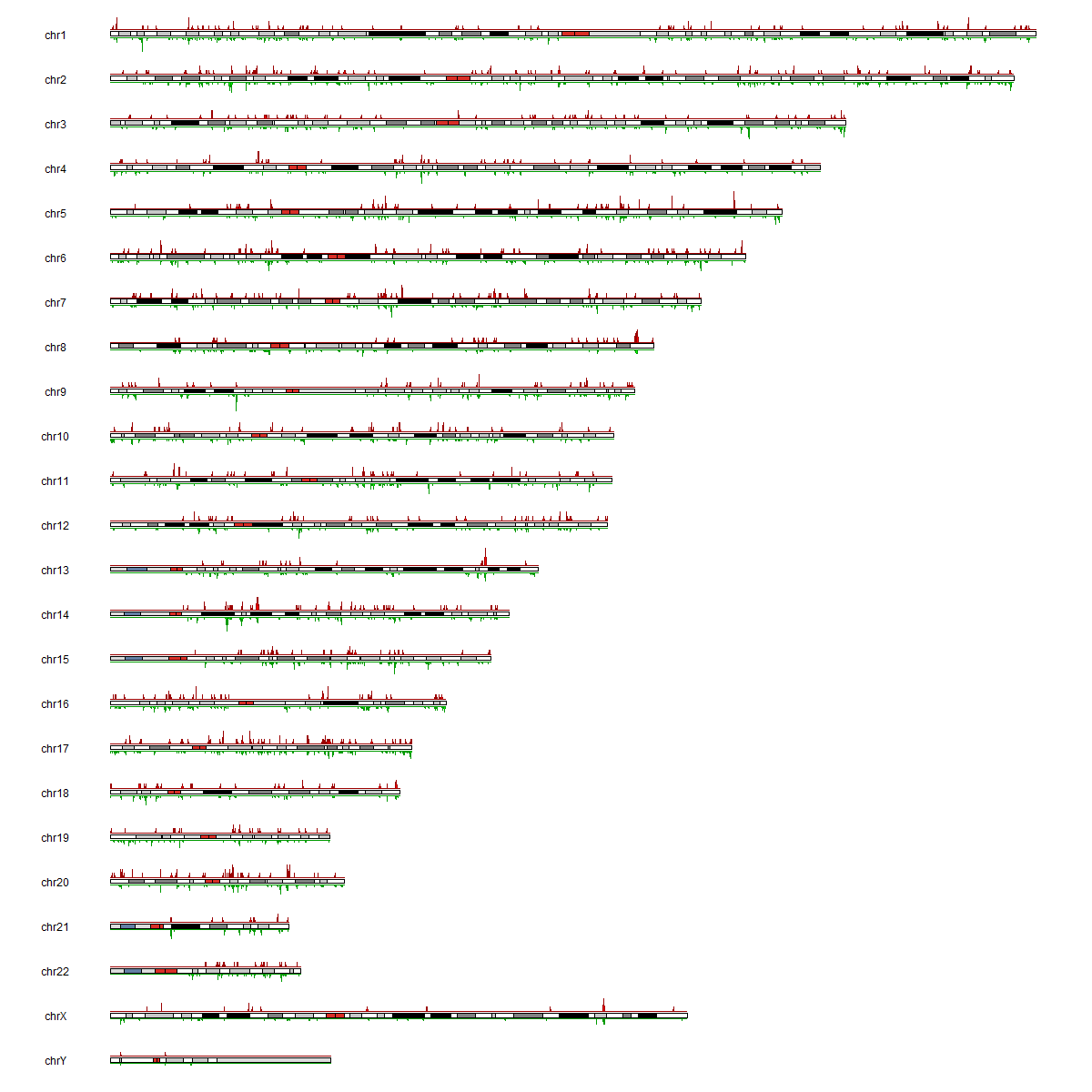
图3. m6A 全基因组分布分析
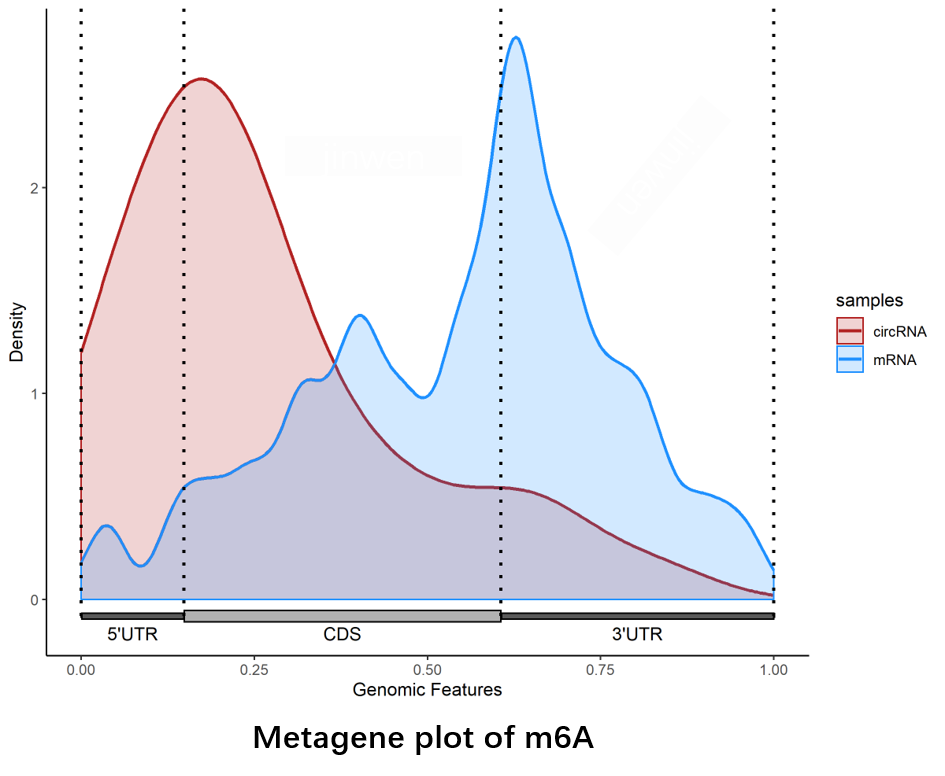
图4. m6A的元基因分析
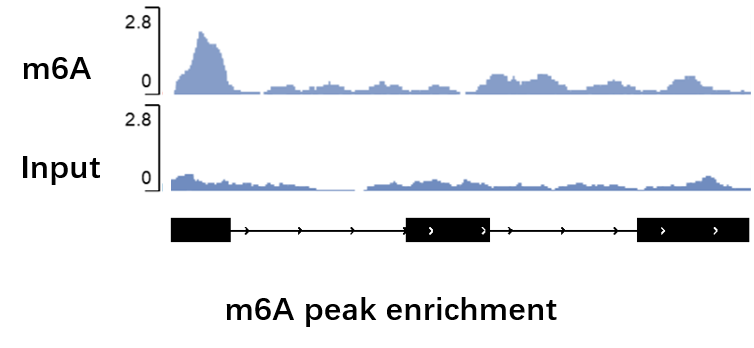
图5. m6A 特定基因丰度展示
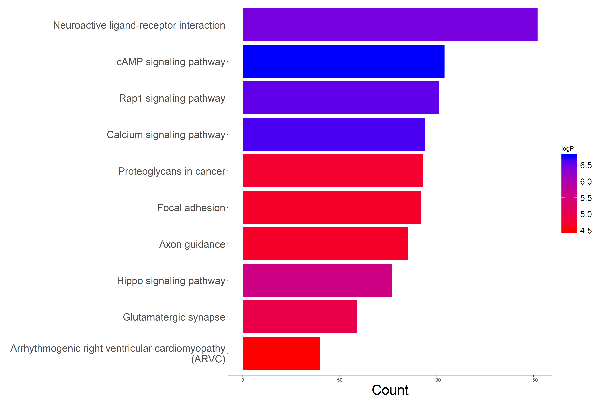
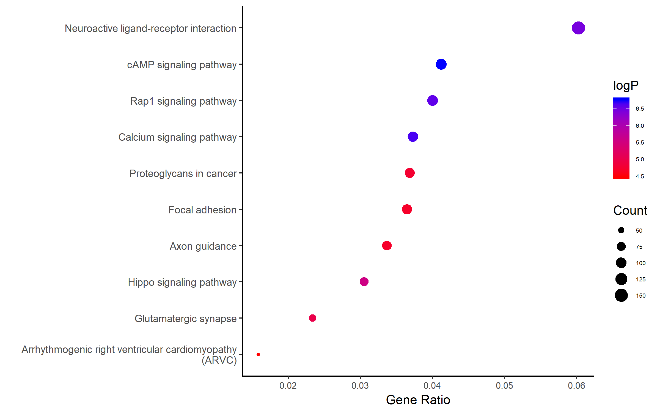
图6. KEGG通路富集分析
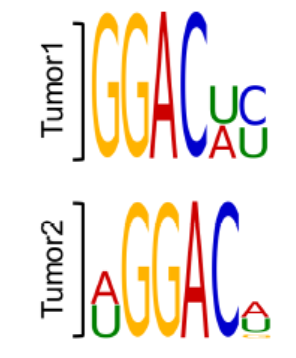
图7. Motif分析
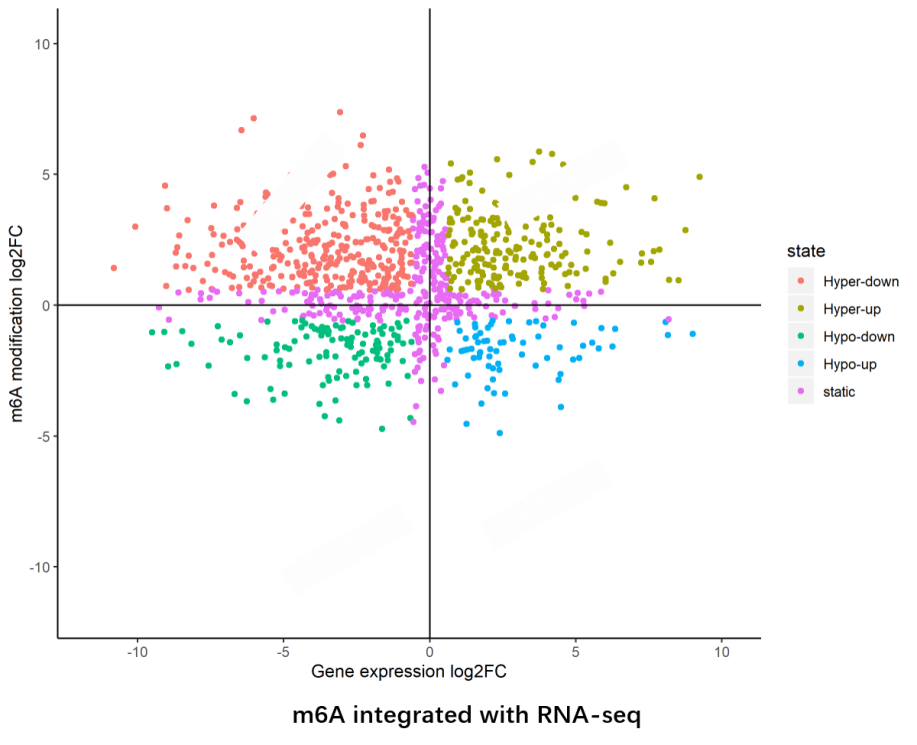
图8. 转录组联合分析四象限图
Table.MeRIP-Seq原始样本送样建议
送样类型
送样量
备注
细胞 (细胞数)
≥5*10^7
无支原体污染
动物组织
≥300mg
植物组织
≥300mg
Table.MeRIP-Seq RNA样本送样建议
送样类型
送样量
完整性(RIN值)
浓度
纯度
total RNA
≥200μg
≥7
≥100ng/μl
无DNA,蛋白/盐离子等污染,样本无色透明不粘稠
*更具体的送样方法请详询销售或技术支持
物种范围:人、小鼠、大鼠等哺乳动物,其他物种请咨询销售或技术支持
Q1:做MeRIP-seq是否还需要另外做普通的转录组测序?
A1:不需要,Input数据即可作为普通全转录组测序数据进行分析。
Q2:为什么MeRIP-seq需要同时测Input和IP样本?
A2:MeRIP-seq实验设置中Input和IP组成一对样品。实验环节IP样品用m6A抗体特异性富集甲基化修饰的RNA片段,而Input仅仅是片段化的RNA则作为对照消减背景噪音,在建库、测序平行开展。结合峰检测分析需整合两个样本的数据,并利用Input数据排除本底表达水平高或非特异性结合的peaks,以提高calling peak的准确性。
Q3:开展MeRIP-seq是否有物种限制?
A3:如不是人、小鼠和大鼠的物种建议先进行咨询。一般有参考基因组,且基因组拼接至染色体水平,gtf注释文件较完整的物种都可以开展;真核、原核或病毒在结合峰检测涉及的软件和参数存在差别,原核或病毒项目建议先评估。
填写需求描述给我们
工具快速咨询
400-8989-400
geneseed@geneseed.com.cn

 购物车
购物车




 广州市黄埔区开源大道11号科技企业加速器A区6栋2楼
广州市黄埔区开源大道11号科技企业加速器A区6栋2楼


 geneseed@geneseed.com.cn
geneseed@geneseed.com.cn