- ·
Ribo-seq
- 产品介绍
- 案例解析
- 结果展示
- 送样建议
服务介绍
广义的翻译组指直接参与翻译过程的所有元件,包括但不限于核糖体、正在翻译的mRNA、调控性RNA、新生肽链及各种翻译因子等;狭义的翻译组特指正在翻译的mRNA。Ribo-seq是指对被核糖体保护的30nt左右的mRNA片段进行深度测序分析,是目前研究翻译组学的主要技术手段之一。
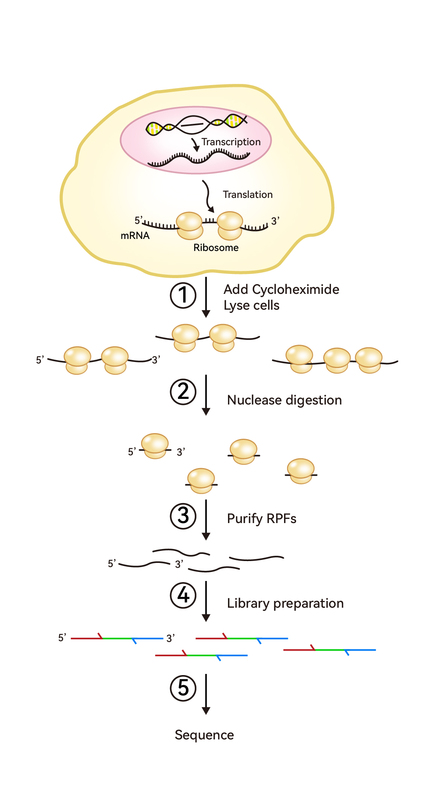
核糖体印迹测序技术路线
测序方案
测序平台:Illumina Novaseq X Plus
测序模式:SE150
测序数据量:30 M raw reads人类心脏翻译组学研究
The Translational Landscape of the Human Heart
Cell (2019). DOI: 10.1016/j.cell.2019.05.010(IF:36.216)
测序策略:Ribo-seq & mRNA-seq
样本分组:80例心脏组织,65例心肌病,15例正常对照
本研究对65例扩张性心肌病(DCM)患者左心室心肌组织和15例正常对照者的左心室心肌组织进行mRNA测序(mRNA-seq)及核糖体印迹测序(ribo-seq),分析了DCM与健康对照组被翻译的RNA分子总体情况,结合之前一项心脏组织蛋白质组学的数据,系统分析了左心室心肌组织中被翻译的ORF和对应的蛋白或多肽的总体信息。共发现1090个uORF、 20763个传统的ORF以及74个dORF。还包括了来自于169个lncRNA 的339个sORF(图1)。此外,本文通过分析ribo-seq数据中被核糖体捕获的circRNA接口序列找到了40个可翻译的circRNA分子。作者分析接口位置Reads的条件是不少于9nt的跨接口碱基,在不少于3个样本且总Reads数不少于5个的才算有效的可翻译circRNA(图2)。这40个circRNA来自39个基因,其中有6个circRNA的翻译产物在早期的蛋白质组学研究中有对应的质谱线索。40个circRNA中也不乏大家耳熟能详的明星分子,包括CDR1as等等。其中circCFLAR,circSLC8A1,circMYBPC3和circRYR2是首次发现的心肌中可被翻译的circRNA分子。
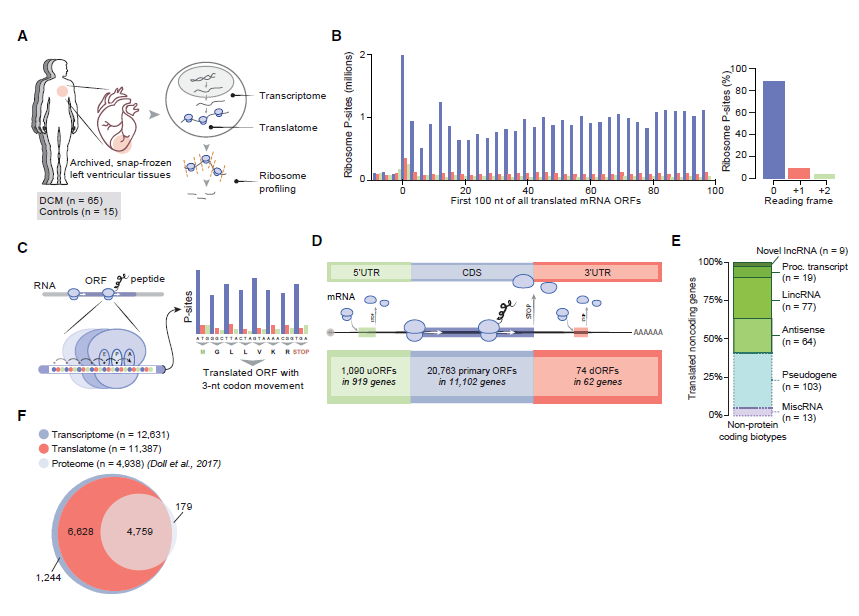
Figure1. 心脏翻译组学研究概览
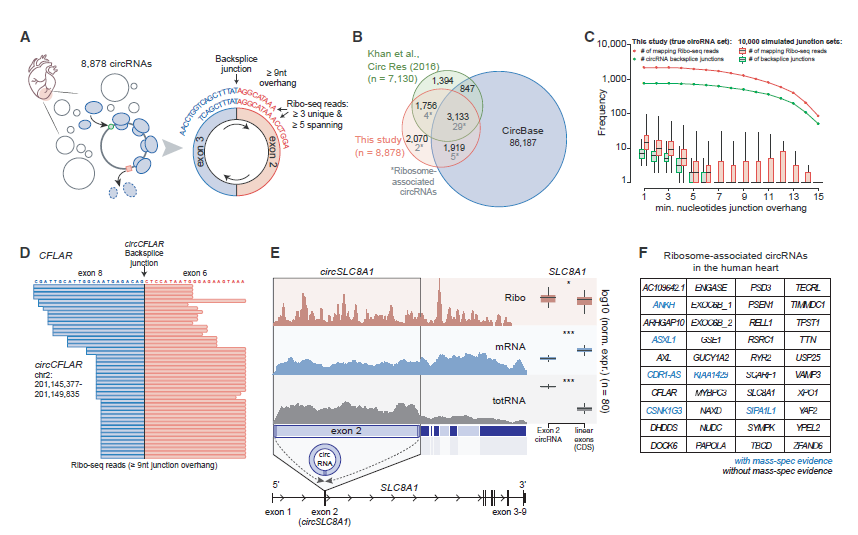
Figure2. Ribo profiling分析可翻译的circRNA分子
生物信息分析
基础分析
原始数据质控检查
比对结果质控
reads 在基因组上的分布分析
基因覆盖度分析
翻译图谱刻画:P-site定义确认
翻译图谱刻画:ORFs统计与对应的氨基酸序列分析
高级分析
翻译组差异基因表达分析
翻译组差异基因GO功能分析
翻译组差异基因KEGG通路分析
翻译组差异基因Rectome通路分析
circRNA翻译事件识别分析
翻译组差异circRNA宿主基因GO功能分析
翻译组差异circRNA宿主基因KEGG通路分析
翻译组差异circRNA宿主基因Rectome通路分析
翻译组差异circRNA的靶向miRNA预测分析
翻译组差异circRNA及靶向miRNA网络调控制图
差异circRNA翻译潜能预测分析
个性化分析
TE翻译效率分析
差异TE基因表达分析
差异TE基因聚类分析
差异TE基因GO/KEGG富集分析
部分结果示例
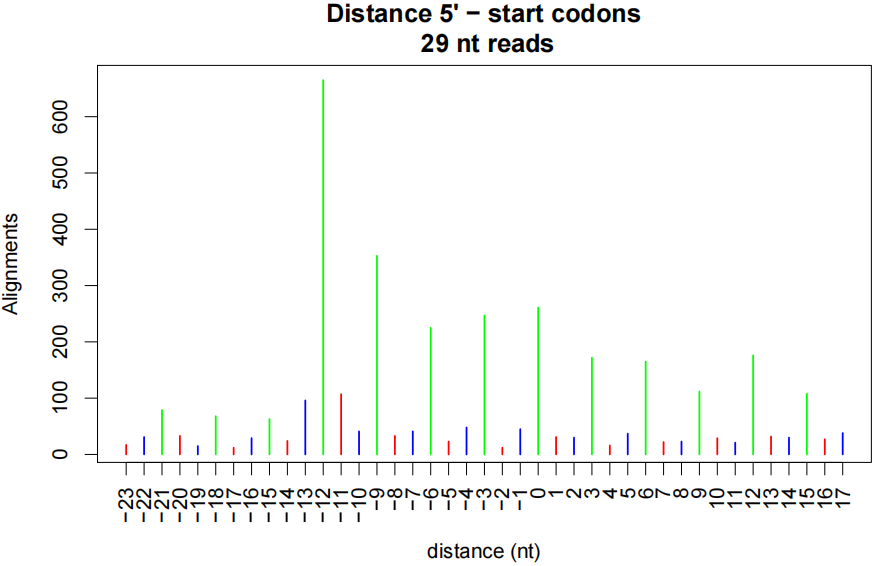
图1. P-site 评估样图
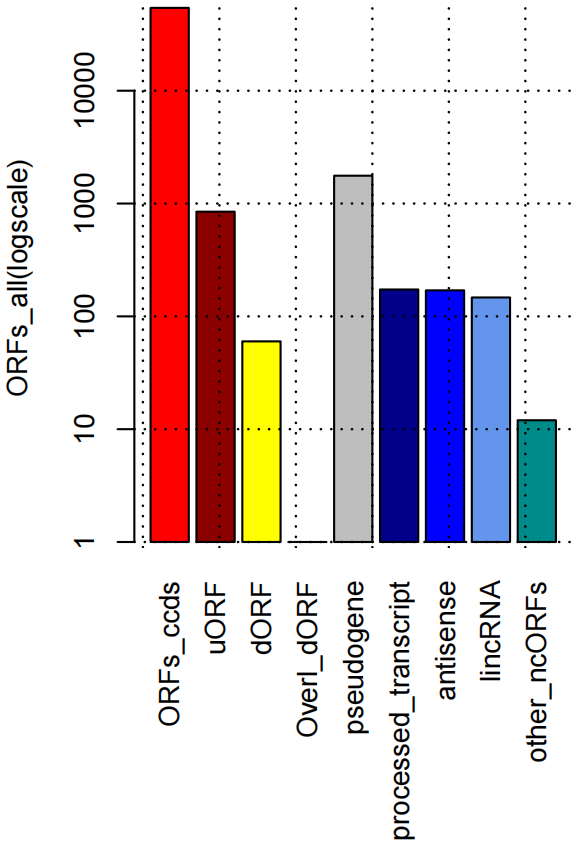
图2. ORF类型数量统计图
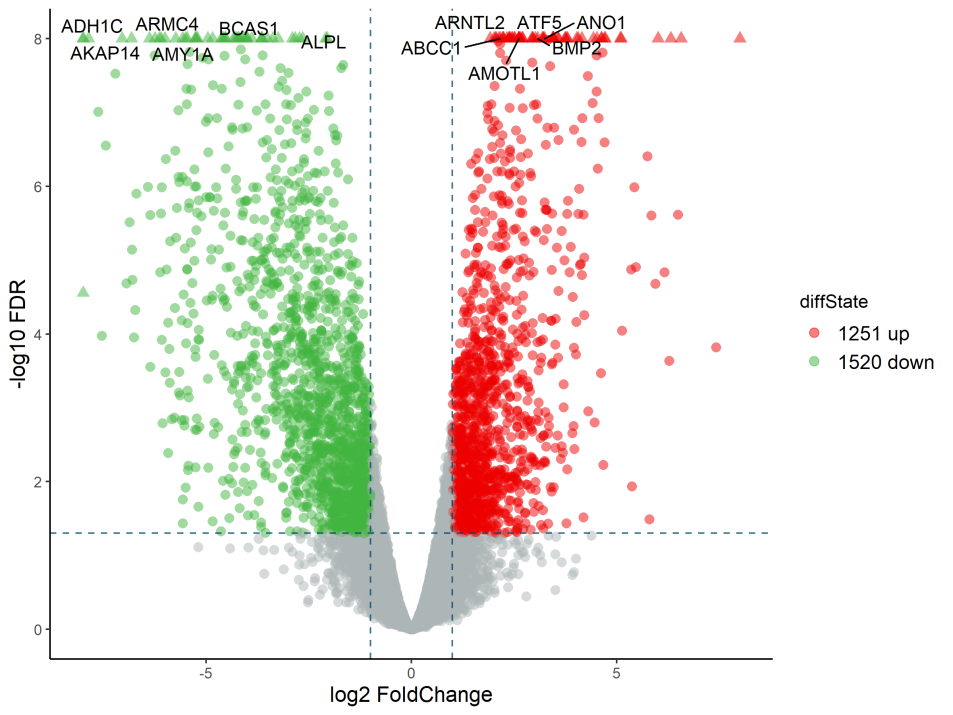
图3. Riboseq差异表达基因的火山图
Table1. Ribo-seq原始样本送样建议
送样类型
送样量
备注
细胞(细胞数)
≥1*10^7
无支原体污染
动物组织
≥200mg
植物组织 ≥200mg 细菌样本
≥200mg
*翻译组测序的细胞样本处理方法和转录组测序不一样,具体处理、保存和运输方法请咨询技术支持。
物种范围:人、小鼠、大鼠,其他物种请详询销售或技术支持
填写需求描述给我们
工具快速咨询
400-8989-400
geneseed@geneseed.com.cn

 购物车
购物车




 广州市黄埔区开源大道11号科技企业加速器A区6栋2楼
广州市黄埔区开源大道11号科技企业加速器A区6栋2楼


 geneseed@geneseed.com.cn
geneseed@geneseed.com.cn