- ·
Nanopore 16S rDNA扩增子测序
- 产品介绍
- 案例解析
- 送样建议
服务介绍
利用特定的16S引物通过对特定环境中的微生物(细菌)DNA特定区域全长16S(V1-V9)进行扩增测序,获取更多的高可变区信息以了解样本中的物种组成以及相对丰度等,显著提高物种注释的分辨率和准确性,更全面的反映微生物的群落结构。以研究微生物群落组成、物种丰度以及样本间群落组成差异情况。
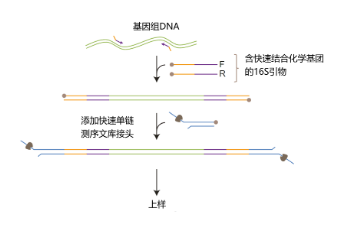
Nanopore 16S rDNA扩增子测序流程图
测序方案
测序模式:Nanopore Sequencing
测序数据量:按需
技术优势
1. 可实现全长16S rDNA的测序,获取更多的高可变区信息,提高物种注释的分辨率和准确性;
2. 可全面准确地展现微生物群落结构。
Zhang T, Li H, Ma S, et al. The Newest Oxford Nanopore R10.4.1 Full-length 16S rRNA Sequencing Enables the Accurate Resolution of Species-Level Microbial Community Profiling. Appl Environ Microbiol. 2023
本研究应用Oxford Nanopore PromethION平台(R9.4.1和R10.4.1)、Pacbio Sequel II和Novaseq平台通过16S rRNA扩增子测序对3个模拟群落和3个环境样本进行测序。模拟群落包括商业模拟群落Zymo D6305、实验室合成群落S1(丰度差异较大)和S2(丰度差异较小),包含实验室合成群落包含了2对同属物种,其中还有具有高GC基因组含量的链霉菌。针对模拟群落,从reads层面评估错误分布、序列一致性分布和正确分类的reads比例;从种属层面比较了不同方法获得的丰度、召回率、精确度、L1距离等指标。针对环境样品,在种属水平上比较了top15丰度、相关性、分类比例等指标;比较了不同平台获得的alpha多样性和beta多样性结果。
1、nanopore平台可检测到极低丰度的物种
在Zymo群落和S2群落的种水平分析中,ONT R10.4.1和PB测序数据都可以获得完整的召回率。针对复杂的S1群落,仅ONT数据获得完整的召回率,其表现更为优秀,且R10.4.1获得了比R9.4.1更小的L1距离。
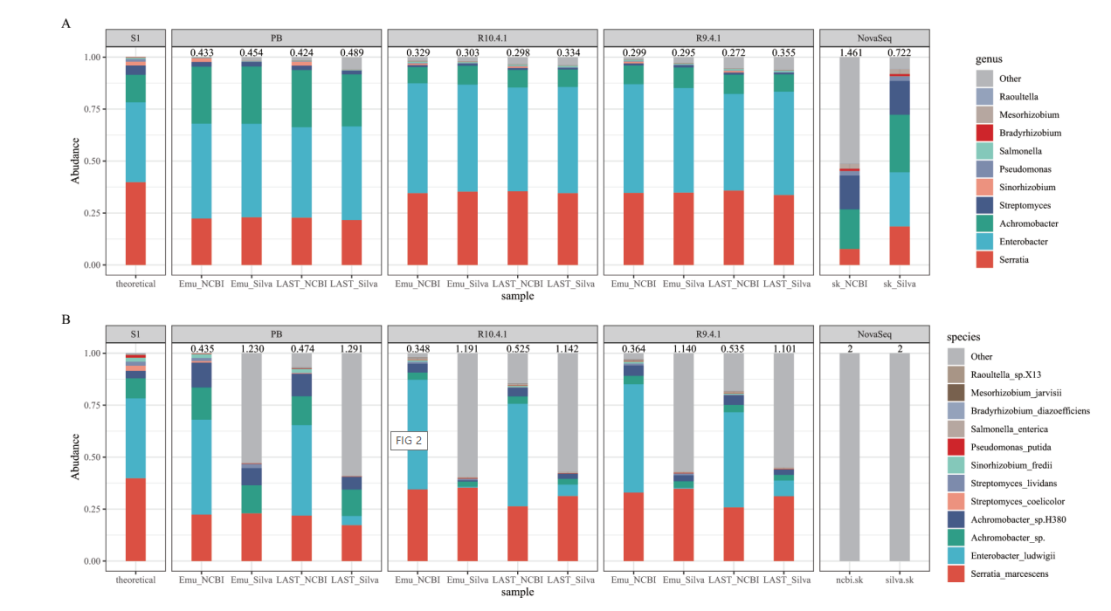
图1:S1样品不同平台基准测试结果
2、nanopore可以检测到更多的种和属分类
在真实环境样本的测试中,ONT R10.4.1数据鉴定到的种和属的数量均多于PB数。在环境样品中R10.4.1数据和PB数据检测到共有种和共有属数目通常最多。在土壤样本中,使用LAST分类器比对NCBI数据库时,二者种水平的相关性可达0.990(p<0.05),二者具有接近Shannon指数和Simpson指数。
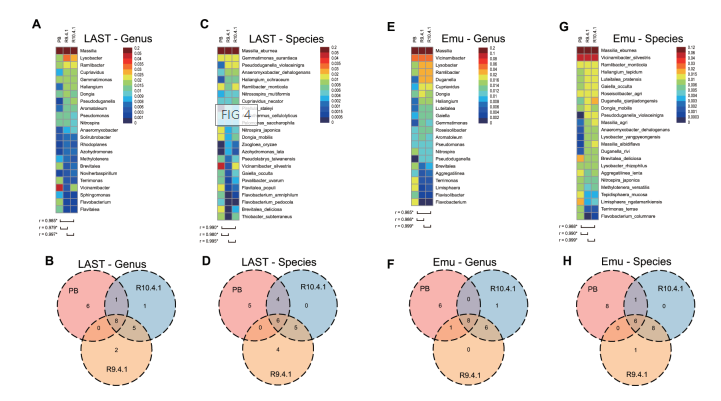
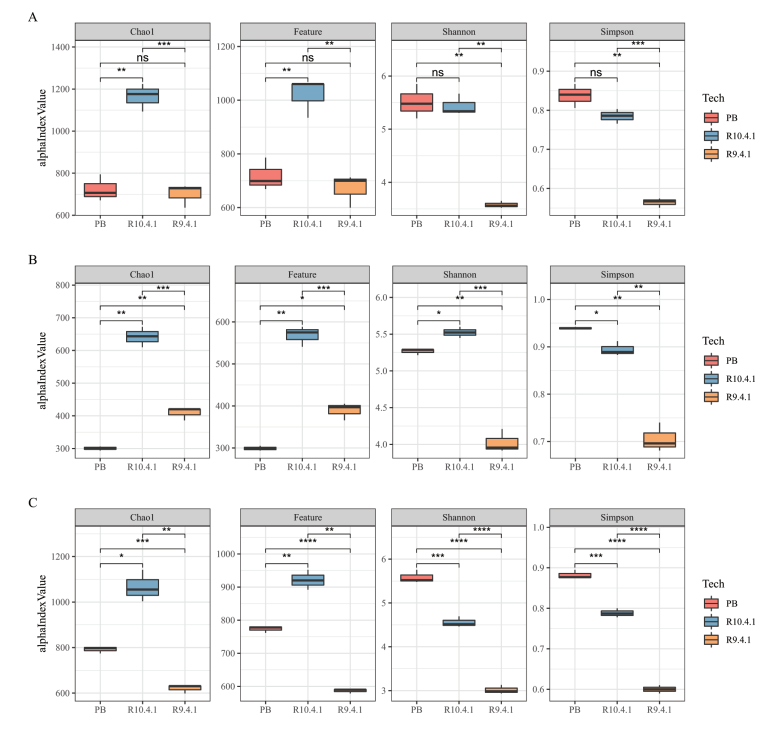
图2:相关性及多样性分析
详询技术支持或销售。
填写需求描述给我们
工具快速咨询
400-8989-400
geneseed@geneseed.com.cn

 购物车
购物车




 广州市黄埔区开源大道11号科技企业加速器A区6栋2楼
广州市黄埔区开源大道11号科技企业加速器A区6栋2楼


 geneseed@geneseed.com.cn
geneseed@geneseed.com.cn