- ·
Nanopore Direct RNA测序
- 产品介绍
- 案例解析
- 结果展示
- 送样建议
服务介绍
Nanopore直接RNA测序技术是对天然RNA进行测序,是对单个RNA分子进行单分子水平的全长测序,能够保留并检测RNA碱基修饰信息,对poly(A)尾长进行相对准确的估算,同时进行全长异构体分析,还原真实RNA特征。
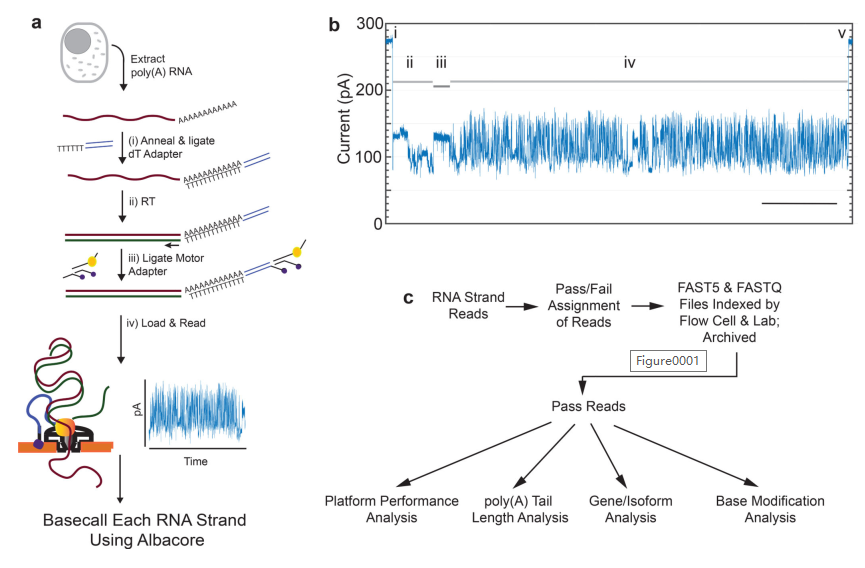
Nanopore Direct RNA测序流程图(2019 Workman et al)
测序方案
测序模式:Nanopore Sequencing
测序数据量:6Gb/样,or 10Gb/样,or 15Gb/样
技术优势
1、Direct RNA技术无需PCR,无GC偏好性提供无偏全长且链特异的RNA序列;
2、Direct RNA技术准确检测poly(A)尾长度为分析RNA稳定性和翻译效率等转录后调控研究提供帮助;
3、Direct RNA技术直接识别RNA碱基修饰信号实现转录和表观信息的一举多得。
利用Nanopore测序技术对人类poly(A)转录组进行直接RNA测序
高通量cDNA测序使我们对转录组的复杂性和调控有了更深入的了解,但是该方法不能完全展现转录组的真实信息,其测序读长较短并且去除了天然RNA的碱基修饰信息。本研究利用Nanopore直接RNA测序技术从人类B淋巴细胞系GM12878分离并测序了原始poly(A) RNA,共生成约990万条通过质控的poly(A) RNA序列,读长N50约1,334bp。利用以上长读长数据,研究者首先检测和分析了转录异构体,在代表10,793个基因的33,984个异构体中,发现52.6%未经注释的剪接连接点,识别出了上千个目前在GENCODE v27中未经注释的基因,并发现了使用短读长测序则无法检测到的等位基因特异性异构体。例如:IFIH1基因,其父系同型保留了8号外显子,而母系同型则不保留8号外显子(图1)。
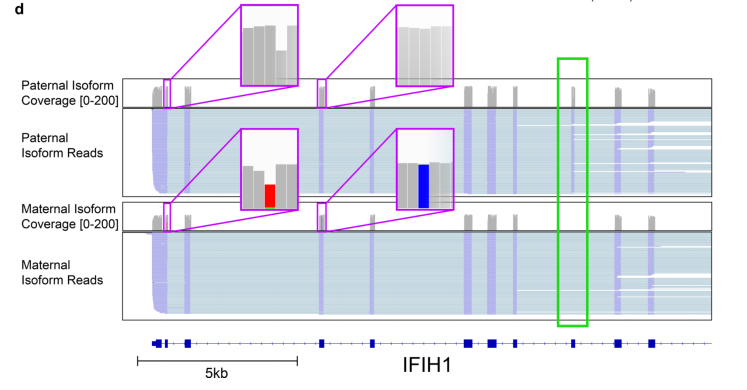
图1 ONT直接RNA测序转录本识别FIH1的等位基因特异性异构体的IGV视图。紫色方框指示等位基因特异性的SNP的位置(灰色为参考,红色和蓝色为SNPs),绿色方框指示可变剪接外显子。
随后结合短读长数据,研究团队直接测量了poly(A)尾长,分析发现了异构体间poly(A)尾的区别。采用ONT直接RNA测序对RNA结合蛋白DEAD-box解旋酶5(DDX5)的转录本poly(A)尾长度进行估计,内含子保留异构体的poly(A)尾长中值为327nt,而其蛋白编码异构体的poly(A)尾长中值为125nt(图2)。
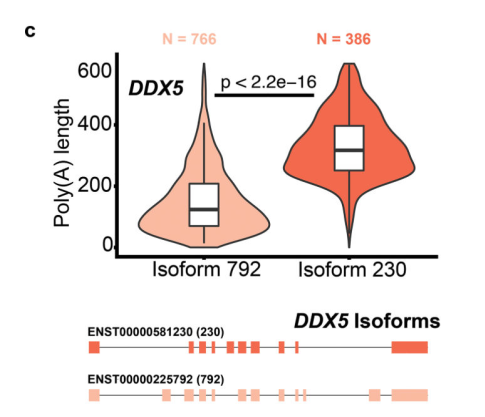
图2 DDX5基因两种转录本异构体的poly(A)尾长分布及基因模型
最后,利用直接RNA测序数据中包含的RNA修饰信息,研究人员筛选了GENCODE敏感亚型的离子电流在GGACU模体(motif)上的改变,发现了86个基因(198个异构体) 的电流变化可以归因于异构体特异性m6A修饰。
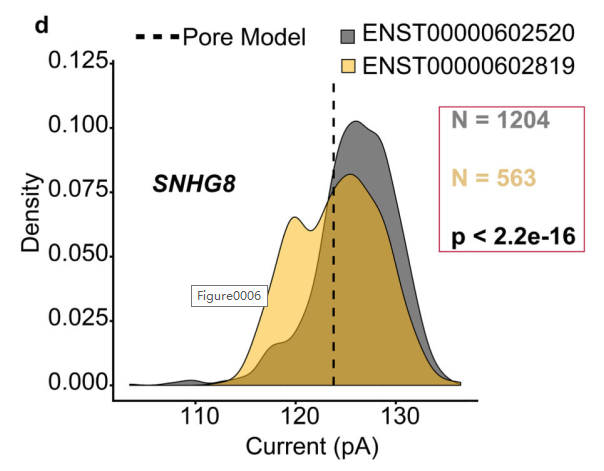
图3 SNHG8基因异构体内GGACU motif的离子电流分布
研究人员表示基于短读长测序的RNA修饰分析去除了修饰之间以及修饰与其他RNA之间的特征,而Nanopore直接RNA测序则具备检测这些远程互作的能力。
参考文献
Workman R E, Tang A D, Tang P S, et al. Nanopore native RNA sequencing of a human poly (A) transcriptome[J]. Nature methods, 2019: 1-9.
生信分析
isoform分析
可变剪切分析
融合基因鉴定
PolyA分析
表达定量
转录本定量
转录本差异分析
富集分析
蛋白质互作网络
甲基化修饰分析
m6A位点注释
甲基化修饰富集分析
m6A差异分析
isoform联合定量表达分析
AltTP分析
功能多样性分析(FDA)
差异富集分析
新生mRNA分析
识别新生mRNA
新生mRNA半衰期分析
mRNA稳定相关性分析
新生mRNA差异分析
Table. Nanopore Direct RNA Sequencing样本送样建议
送样类型
送样量
完整性(RIN值)
浓度
纯度
Total RNA(组织、细胞、全血等)
≥50μg
≥8
≥180ng/μL
无DNA,蛋白/盐离子等污染,样本无色透明不粘稠
RNA带poly(A)尾
≥300ng
-
≥50ng/μL
无DNA,蛋白/盐离子等污染,样本无色透明不粘稠
*更具体的送样方法请详询销售或技术支持
物种范围
人、小鼠、大鼠等哺乳动物,植物等其他物种详询销售或技术支持
填写需求描述给我们
工具快速咨询
400-8989-400
geneseed@geneseed.com.cn

 购物车
购物车




 广州市黄埔区开源大道11号科技企业加速器A区6栋2楼
广州市黄埔区开源大道11号科技企业加速器A区6栋2楼


 geneseed@geneseed.com.cn
geneseed@geneseed.com.cn